#trendsinbiochemicalsciences #cellule #protéine #enzyme #substrat #protéolysemembranaire Comprendre la protéolyse membranaire : de la dynamique des protéines à la cinétique de réaction
Publié le 05 mai 2015 par Tartempion77
@NZarjevski
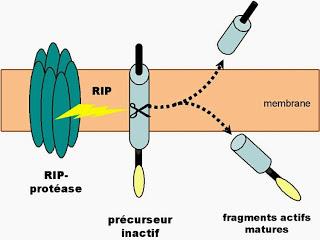
Schéma général de la protéolyse intramembranaire régulée (RIP)
Source iconographique et légendaire: http://theses.ulaval.ca/archimede/fichiers/21080/ch01.html
La protéolyse membranaire - à savoir le clivage des protéines au niveau de la surface plane de la membrane - est un phénomène répandu qui contribue à l’activation fonctionnelle des substrats impliqués dans certaines maladies. Bien que différentes familles de protéases intramembranaires aient été découvertes et caractérisées, nous ne savons pas, actuellement, comment ces enzymes font la différence entre substrat et non-substrat, comment s’accomplit le clivage site-spécifique, ou quels sont les facteurs déterminant le taux de protéolyse. En mettant l’accent sur la gamma-secretase (Ƴ - secretase) et les protéases rhomboïdes, nous prétendons que les réponses à ces questions peuvent venir des données expérimentales comme celles des cinétiques de réaction ; ainsi que celles de la détermination des sites de clivage, jusqu’à celles relatives à la structure et la dynamique conformationnelle des substrats et enzymes. D. Langosh et al, dans Trends in Biochemical Sciences, publication en ligne en avant-première, 1ermai 2015Source : Science Direct / Traduction et adaptation : NZ

